Eine neue Studie aus unserem Forschungsprojekt zeigt, dass Pflanzenbeobachtungen, die mit Bestimmungs-Apps gesammelt werden, Aussagen über die Entwicklungsstadien von Pflanzen zulassen – sowohl kleinräumig als auch europaweit. [Studie lesen]
Warum ist die Dokumentation der Phänologie wichtig?
Viele Pflanzen in gemäßigten Klimazonen durchlaufen jedes Jahr einen Ablauf aus Blüte, Blattaustrieb, Fruchtbildung, Blattfärbung und Blattfall. Diesen Prozess nennt man Phänologie, und er und wird stark von lokalen klimatischen Bedingungen beeinflusst (zum Beispiel von der Anzahl der Tage im Jahr, an denen eine bestimmte zum Wachstum notwendige Mindesttemperatur erreicht wird, siehe „Growing Degree Day“ oder GDD). Deswegen ist es nicht verwunderlich, dass sich der Klimawandel stark auf die Phänologie auswirkt. Beispielsweise beginnt der Frühling mittlerweile früher als noch in den 1950er Jahren, wodurch die Vegetationsperiode viel schneller einsetzt als damals. Solche Veränderungen haben Auswirkungen auf landwirtschaftliche Abläufe und können außerdem auch zu ökologischen Ungleichgewichten führen: Pflanzen beginnen beispielsweise zu blühen, noch bevor ihre Bestäuber aktiv sind. Aber nicht alle Pflanzen reagieren gleichermaßen auf klimatische Veränderungen. Arten, die ein größeres Toleranzspektrum für warme Tage haben, oder bei denen andere Faktoren die Phänologie bestimmen, bleiben von Verschiebungen nahezu unberücksichtigt. Um ein wirklich präzises Verständnis für den Einfluss des Klimas auf die Pflanzenphänologie zu erlangen, ist es wichtig, die Phänologie von möglichst vielen verschiedenen Arten, in unterschiedlichen Ländern und geografischen Regionen zu dokumentieren.
Wie funktioniert das phänologische Monitoring?
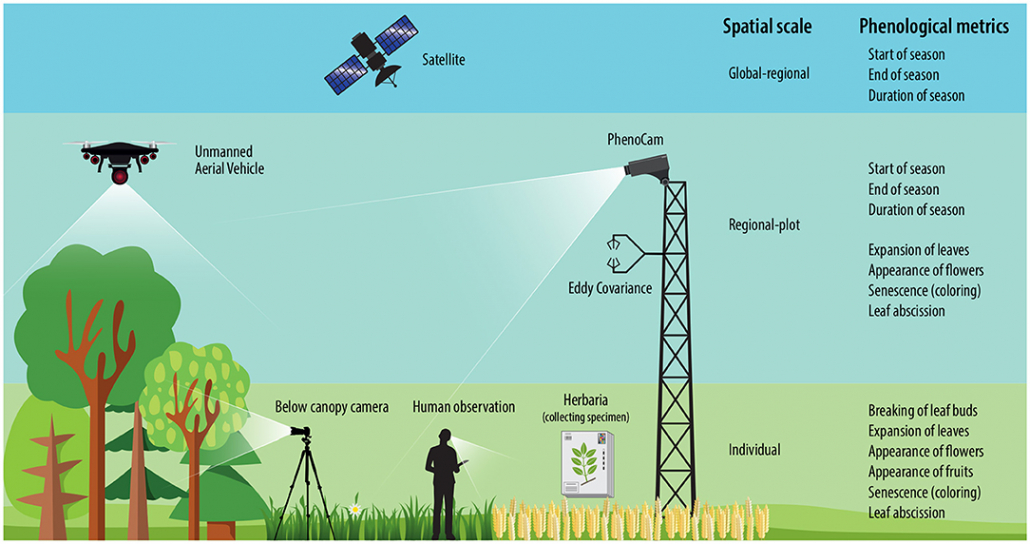
Phänologie wird heute bereits über verschiedene Methoden dokumentiert. Satellitenbilder erkennen das Ergrünen ganzer Landstriche, Kameras in Baumkronen fertigen automatisierte Bilderserien über den Zustand der darunter liegenden Vegetationsschicht an. Solche Datensätze erlauben Aussagen über große Skalen, lassen aber kaum Rückschlüsse auf die Phänologie von einzelnen Arten oder gar Individuen zu. Hierfür gibt es Initiativen, die mit Hilfe von geschulten Freiwilligen durchgeführt werden. Die Zahl dieser Bürgerwissenschaftler:innen geht jedoch immer weiter zurück, und zudem ist diese Art der Datenerhebung in der Regel auf bestimmte Pflanzenarten (oftmals Bäume), Länder oder noch kleinere Regionen beschränkt.
Kann man mit Flora Incognita Phänologie dokumentieren?
Daten, die über Pflanzenbestimmungs-Apps wie Flora Incognita erhoben werden, können hier eine Lösung sein. Das haben die Wissenschaftler:innen unseres Projekts bereits 2023 nachgewiesen: Pflanzen werden vor allem dann wahrgenommen und fotografiert, wenn sie einerseits auffällig sind und zudem blühen, bunte Früchte tragen oder Herbstlaub. So entstehen Beobachtungsmuster, die phänologische Events anzeigen. Diese Muster decken sich in vielen Fällen mit denen, die der Deutsche Wetterdienst (DWD) in Bezug auf den Blühbeginn von Arten in Deutschland veröffentlicht. Angenommen, der DWD registriert in einem Jahr einen früheren Blühbeginn des Holunders als im Vorjahr, dann spiegelt sich diese Verschiebung auch in den Bestimmungsanfragen von Flora Incognita wider.
Details zu dieser Studie findest Du in diesem Artikel: Phänologie-Monitoring mit Flora-Incognita-Pflanzenbeobachtungen.
Neue Studie zeigt Phänologien und bioklimatische Zusammenhänge über ganz Europa
Unsere neue Publikation zeigt nun, dass Smartphone-Beobachtungen sogar bekannte überregionale phänologische Muster widerspiegeln, wie z. B.
- die spätere Blüte vieler Arten in Nord- und Osteuropa oder
- die spätere Blüte vieler Arten in größeren Höhenlagen, aber auch
- eine europaweite Verschiebung des Blühbeginns zwischen den Jahren, wie es bereits für Deutschland nachgewiesen wurde.
Das beweist, dass die von Pflanzenbestimmungs-Apps generierten Daten eine zuverlässige Quelle für das Vorkommen von Pflanzen zu einem bestimmten Zeitpunkt und an einem bestimmten Ort sind und sich gut für die Beantwortung weiterer Forschungsfragen eignen – auch in größeren Maßstäben.
Die Ergebnisse der Studie im Überblick
Pflanzen blühen eher, wenn es mehr warme Tage gibt
Wir haben die europaweiten Beobachtungsdaten (Quellen: Flora Incognita, aber auch Meldeplattformen wie iNaturalist) aus 2020 und 2021 von 20 verschiedenen Pflanzenarten miteinander verglichen. Dabei konnten wir feststellen, dass insbesondere die Frühlingsblüher wie beispielsweise der Gamander-Ehrenpreis Veronica chamaedrys 2020 zeitiger geblüht haben – bis zu zwei Wochen eher als 2021.

Eine Analyse der Temperatur am jeweiligen Standort zeigte auf, dass es im Frühjahr 2020 deutlich mehr Tage gab, an denen im Mittel 5°C oder darüber erreicht wurden, die Pflanzen also in kürzerer Zeit mehr Wärme aufnehmen konnten. Bei Arten, die später im Jahr blühen, wie der Rainfarn Tanacetum vulgare oder der Gewöhnliche Natternkopf Echium vulgare, war der Effekt weniger ausgeprägt.
Pflanzen blühen später, wenn sie in höheren Lagen, weiter im Osten oder im Norden wachsen
Es waren jedoch nicht nur Muster zwischen den Jahren, sondern auch zwischen verschiedenen Regionen zu erkennen. Es ist bekannt, dass die gleiche Art je nach Standort zu unterschiedlichen Zeiten blüht. (-> Hopkins‘ bioklimatisches Gesetz) Wenn beispielsweise die gleiche Pflanzenart in Schweden und Spanien vorkommt, blüht die spanische Pflanze einige Tage oder sogar Wochen früher als die im Norden. Die genaue Anzahl der Tage bis zur Blüte variiert natürlich je nach Pflanzenart. Auch diese Gesetzmäßigkeit lässt sich mit Flora-Incognita-Daten abbilden:

Diese Abbildung zeigt den Median der Beobachtungsdaten für die drei bereits vorgestellten Pflanzenarten. Rosa und orange Farben zeigen an, dass die Art am jeweiligen Ort früh im Jahr blühte, während die gleiche Art an einem anderen Standort später blühte (dunkelgrün und blau codiert). Deutlich setzen sich nicht nur die Längen- und Breitengrade ab, sondern auch die Mittel- und Hochgebirge. Details zu den Daten und angewandten Methoden sind in der Publikation ersichtlich, die am E nde des Artikels verlinkt ist.
Alle untersuchten 20 Pflanzenarten ließen sich in eines von drei Hauptmustern eingliedern, die hier beispielhaft abgebildet sind. Veronica chamaedrys zeigt im Jahr 2020 mehr rötliche Farben als im Jahr 2021; wie bereits erwähnt ist dies auf die wärmeren Temperaturen im Frühjahr 2020 zurückzuführen. Echium vulgare zeigt über die Jahre hinweg nur geringfügige Reaktionen auf unterschiedliche Klimabedingungen, und für Tanacetum vulgare konnten wir feststellen, dass Phänologie im Vergleich zu den anderen Arten ein umgekehrtes Muster aufweist: Rainfarn blüht in östlichen, nördlichen und hohen Lagen eher als seine Geschwister in westlichen, südlichen und niedrig gelegeneren Teilen Europas. Auch dieses Phänomen wurde bereits in der wissenschaftlichen Literatur beschrieben. Arten, die eigentlich viele warme Tage bis zur Blüte brauchen, haben sich an kalte Standorte mit einer Verkürzung der Vegetationszeit und einem zeitigeren Blühtermin angepasst.
Zusammenfassung
Die neue Publikation zeigt zum ersten Mal auf einer europaweiten Skala zeitliche und räumliche Verschiebungen von Pflanzenphänologie anhand von Daten, die nicht gezielt für diesen Zweck gesammelt wurden. Für die Nutzer:innen von Flora Incognita bedeutet das, dass jede einzelne Pflanzenbestimmung mehr als nur die eigene Neugierde befriedigt. Durch die Dokumentation von Pflanzenvorkommen zu einer bestimmten Zeit an einem bestimmten Ort schaffen sie eine wachsende und robuste Datenquelle zur Phänologie, die keine nationale Grenzen kennt, neue Arten mit einschließt und zahlreiche weiterführende Forschungsfragen beantworten kann.
Vielen Dank für Eure Neugier.
Die neue Veröffentlichung ist ab sofort frei verfügbar:
Rzanny, M., Mäder, P., Wittich, H.C. et al. Opportunistic plant observations reveal spatial and temporal gradients in phenology. npj biodivers 3, 5 (2024). https://doi.org/10.1038/s44185-024-00037-7
Veronica chamaedrys im Titelbild: aufgenommen von Ilse Schönfelder.